숄레스키 분해를 이용한 마할라노비스 거리
"현재 인과추론 패키지 개발 업무 중 거리 기반 매칭 기능을 조사하며 얻은 지식을 정리하였습니다 :)
마할라노비스 거리를 구하는 방법에 대해 기술합니다"
마할라노비스 거리
$n$개의 샘플이 있고, 각 샘플은 $k$차원 공간(feature의 수)의 벡터로 표현된다고 합시다
그리고 임의의 두 개의 샘플에 대한 $1 \times k$ 형태의 행 벡터가 $\vec{v} = (v_1, v_2, ..., v_{k-1}, v_k)$과 $\vec{u} = (u_1, u_2, ..., u_{k-1}, u_k)$일 때 마할라노비스 거리의 정의는 아래와 같습니다
$$ mahalanobis(\vec{v}, \vec{u}) = \sqrt{(\vec{v} - \vec{u}) {\sum}^{-1} (\vec{v} - \vec{u})^T} $$
여기서 ${\sum}$는 $k \times k$ 형태의 feature 공분산 행렬(covariance matrix)입니다. 각 feature간 퍼짐의 정도를 나타냅니다.
마할라노비스 거리 계산 시 중간에 공분산 행렬의 역행렬 ${\sum}^{-1}$ 가 들어간 이유는 feature의 퍼짐이 클수록 거리 계산 시 down scaling하기 위해서 입니다. 샘플의 feature 간 거리가 동일하여도 넓은 값에 퍼져 있는 feature의 경우 그렇지 않은 feature보다 거리를 작게 보정해준다는 느낌으로 이해하면 좋습니다.
본 포스팅은 숄레스키 분해를 이용한 마할라노비스를 구하는 방법에 초점을 맞추어 작성하였으므로, 마할라노비스 거리에 대한 개념은 이정도로 마무리하겠습니다. 상세한 내용은 해당 포스팅👏에 잘 설명되어 있으니 참고하시면 좋습니다
숄레스키 분해
$k \times k$ 정사각행렬 $A$가 있을 때, 이를 하삼각행렬 $L$, 상삼각행렬 $U$로 분해하는 것을 LU 분해라고 합니다. 일반적으로 $L \neq U^T$ 입니다
$$ A = LU $$
그리고 LU 분해의 특수한 형태가 숄레스키 분해입니다. 숄레스키 분해에서 $L = U^T$이고, 따라서 아래와 같습니다
$$ A = L L^T $$
갑자기 숄레스키 분해에 대해 왜 언급하는가? 싶으실 수 있습니다. 마할라노비스 거리 계산 시 숄레스키 분해를 활용하면 계산 효율성을 높일 수 있기 때문입니다
숄레스키 분해를 이용한 마할라노비스 거리 구하기
개념
위에서 사용한 공분산 행렬의 역행렬 ${\sum}^{-1}$ 는 $k \times k$ 형태의 정사각행렬이므로 아래와 같이 숄레스키 분해 할 수 있습니다
$$ {\sum}^{-1} = L L^T $$
이를 대입하여 $\vec{v}$와 $\vec{u}$ 간의 마할라노비스 거리를 작성하면 아래와 같습니다
$$ mahalanobis(\vec{v}, \vec{u}) = \sqrt{(\vec{v} - \vec{u}) {\sum}^{-1} (\vec{v} - \vec{u})^T} = \sqrt{(\vec{v} - \vec{u}) L L^T (\vec{v} - \vec{u})^T}$$
여기서 $X = (\vec{v} - \vec{u}) L$ 라고 한다면 $X^T = L^T (\vec{v} - \vec{u})^T$ 가 됩니다. 즉, 아래와 같이 전개됩니다.
$$ mahalanobis(\vec{v}, \vec{u}) = \sqrt{(\vec{v} - \vec{u}) L L^T (\vec{v} - \vec{u})^T} = \sqrt{X X^T}$$
$X$에 L2 norm을 취한 후 square root를 씌운 형태입니다. 이는 유클리디안 거리를 구하는 방법입니다.
즉, 원 데이터에 특정 처리를 한 후, 그 데이터를 이용하여 샘플 간 유클리디안 거리를 구하면 됩니다
정리하면, 숄레스키 분해를 이용하여 샘플 간 마할라노비스 거리를 구하는 방법은 아래와 같습니다
- 원 데이터 ($n \times k$)의 공분산 행렬의 역행렬 ${\sum}^{-1}$ ($k \times k$)을 숄레스키 분해하여 하삼각행렬 $L$ ($k \times k$)을 구합니다
- 원 데이터에 하삼각행렬 $L$을 행렬 곱 취하여 행렬 $X$ ($n \times k$)를 구합니다
- 행렬 $X$의 샘플 간 유클리디안 거리를 구한 것이 원 데이터의 샘플 간 마할라노비스 거리를 구한 것과 같습니다
코드를 통한 확인
iris 데이터를 이용하여 숄레스키 분해를 이용한 마할라노비스 거리에 대해 살펴보겠습니다
150개의 샘플과 4개의 feature (sepal_length, sepal_witdh, petal_length, petal_width)를 사용합니다
데이터 로딩
import pandas as pd
import numpy as np
from sklearn import datasets
from sklearn.metrics import pairwise_distances
iris_dict = datasets.load_iris()
iris = pd.DataFrame(iris_dict['data'], columns=iris_dict['feature_names'])
iris['Species'] = iris_dict['target_names'][iris_dict['target']]
feat = iris.drop('Species', axis=1) # samples * features dataframe숄레스키 분해 - 하삼각행렬 $L$ 반환
cov = np.array(feat.cov()) # 공분산 행렬
cov_inv = np.linalg.inv(cov) # 공분산 행렬의 역행렬
cov_inv_L = np.linalg.cholesky(cov_inv) # cholesky 분해 - 하삼각행렬
print(cov_inv_L)
참고) 하삼각 행렬 곱을 통한 원본 행렬과 비교 : ${\sum}^{-1} = L L^T$
# 숄레스키 분해 확인 : 공분산 행렬 역행렬 = 하삼각행렬 * 하삼각행렬^T
print(cov_inv) # 공분산 행렬 역행렬
print(np.matmul(cov_inv_L, cov_inv_L.T)) # 하삼각 행렬 * 하삼각행렬^T
마할라노비스 거리 구하기 - 기본 방법
# 기본 마할라노비스 거리 계산 방법으로 계산
# VI : 공분산행렬 역행렬 - 마할라노비스 거리 계산 시 필요함
mah1 = pairwise_distances(feat, feat, metric='mahalanobis', VI=cov_inv)
print(mah1)
마할라노비스 거리 구하기 - 숄레스키 분해 사용
위에서 기본 방법으로 구한 마할라노비스 거리와 결과가 같은 것을 알 수 있습니다 :)
X = np.matmul(feat, cov_inv_L) # 원 데이터 * 하삼각행렬
mah2 = pairwise_distances(X, X, metric='euclidean') # euclidean 방법으로 계산
print(mah2)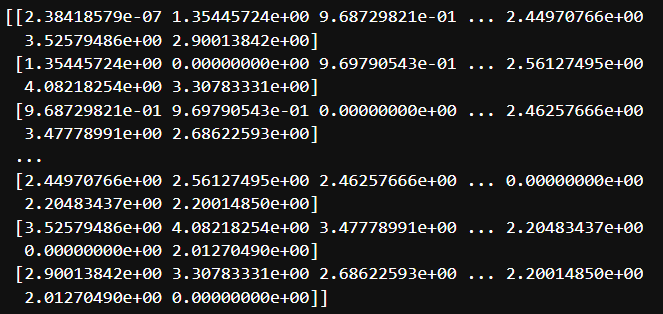
References
- 마할라노비스 거리 개념
- 숄레스키 분해를 이용한 마할라노비스 거리 구하기
마치며
행렬 분해를 이용하여 마할라노비스 거리를 구하는 방법에 대해 알아보았습니다.
읽어주셔서 감사합니다 😉
▼ 글이 도움이 되셨다면 아래 클릭 한번 부탁드립니다 :) ▼